Vous allez être redirigé vers:
Contexte
Les essais cliniques produisent une quantité massive de données. Dans la plupart des cas, le défi réside moins dans le volume de données à traiter que dans leur diversité. Les sources de données sont très nombreuses et ne cessent d'augmenter. On pense d'abord aux données collectées via les formulaires que les médecins doivent remplir, après chaque visite d'un patient sur un site, via un système de capture électronique de données (EDC), ou encore les formulaires de rapport produits par les laboratoires. Mais aujourd'hui, les essais cliniques sont de plus en plus centrés sur le patient, et les résultats électroniques rapportés par le patient (ePRO) sont collectés via des formulaires web ou des téléphones portables. En outre, la quantité de données d'images (scanner, IRM, biopsie...) augmente également ; et le développement de wearables non invasifs collectant des signaux (fréquence cardiaque, niveau d'oxygène dans le sang...) accroît également le besoin de systèmes appropriés de stockage et de manipulation des données.
C'est d'autant plus difficile à mettre en pratique car la diversité des données s'accompagne d'une augmentation des métadonnées, c'est-à-dire des données sur les données, qui doivent être correctement stockées et gérées. Même si de nombreuses organisations font pression en faveur de la normalisation, c'est loin d'être la norme à l'heure actuelle, et les gestionnaires de données doivent souvent regrouper et mettre en correspondance les données dans un format commun pour rendre l'analyse possible.
Ces changements mettent de plus en plus de pression sur les épaules des gestionnaires de données cliniques, l'orfèvre des données cliniques, qui doivent identifier et configurer les technologies pertinentes pour que les données cliniques soient prêtes à être soumises aux autorités et disponibles dans le bon format pour les bio-statisticiens, les médecins et autres investigateurs. En outre, toutes les données cliniques générées au cours d'un essai clinique, le Trial Master File (TMF), doivent être correctement stockées, organisées et mises à disposition. Cela doit être fait pour des raisons réglementaires, mais aussi pour permettre des analyses croisées ou optimiser les opérations de l'essai clinique.
Tendances technologiques
Des services intégrés de mieux en mieux aux plateformes d'essais cliniques.
Les gestionnaires de données cliniques passent une grande partie de leur temps à transférer des données d'une application à une autre. Ces transferts de données augmentent de nos jours, car un portefeuille de services de plus en plus diversifié est nécessaire pour mener correctement un essai clinique. Ces transferts de données sont cependant souvent manuels, ce qui prend du temps et est source d'erreurs. En effet, il n'est pas rare qu'un gestionnaire de données cliniques, pour effectuer certaines transformations de données, doive se connecter à un système EDC déployé sur le cloud A, extraire certaines données d'étude en interrogeant une base de données ou en exportant des fichiers complets, puis se connecter à un autre serveur sur le cloud B pour utiliser des packages SAS ou R paramétrés par des fichiers Excel. C'est bien trop complexe ! Les capacités de transformation des données devraient être intégrées à l'EDC. En outre, les transferts manuels de données empêchent tout traitement des données en temps quasi réel, ce qui est absolument nécessaire, par exemple pour le suivi de la pharmacovigilance.
Pour automatiser/éliminer ces transferts de données manuels, de nombreux fournisseurs combinent différentes applications dans une plateforme intégrée, où différents services sont interconnectés et peuvent fonctionner de manière transparente. Le nombre et le niveau de maturité des services intégrés varient d'un fournisseur à l'autre, mais ces plateformes sont actuellement en plein développement. Certains fournisseurs proposent une couverture complète des services d'essais cliniques : intégration d'un système de gestion des essais cliniques (CTMS), d'un système d'EDC, d'un eTMF et de capacités de reporting avancées ; tandis que d'autres ont commencé par un simple système d'EDC et l'étendent avec des capacités de transformation CDISC-SDTM ou des systèmes de reporting de pharmacovigilance. En outre, avec l'augmentation des données provenant des laboratoires et des dispositifs portables, des outils spécifiques d'ingestion et de transformation des données sont également intégrés.
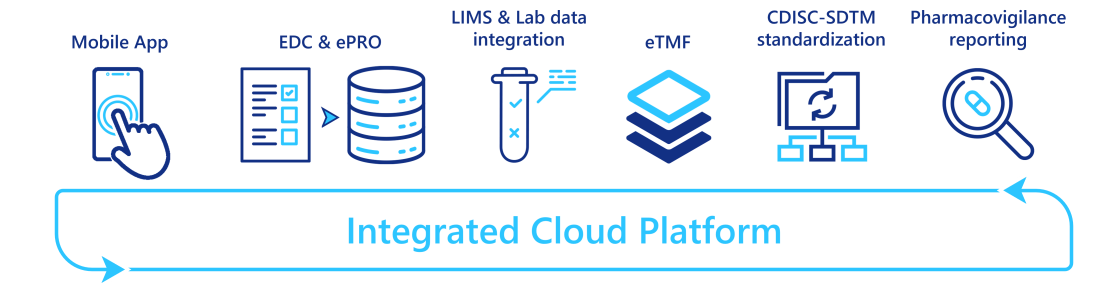
En fournissant un point d'accès unique à une myriade de services, ces platesformes intégrées réduisent les transferts de données fastidieux entre les applications, et libèrent ainsi du temps pour les gestionnaires de données cliniques qui peuvent alors se concentrer sur des tâches plus utiles.
Référentiels de métadonnées cliniques (CMDR)
Les métadonnées sont d'une importance capitale lorsqu'on veut faire interopérer les technologies et promouvoir la réutilisation de code. Elles sont particulièrement importantes pour les gestionnaires de données qui doivent concevoir le flux de transformation des données, depuis la saisie des données par le système EDC jusqu'au format de données CDISC SDTM requis pour la soumission aux autorités. Ce flux se compose de deux tâches principales : la configuration du système EDC sur la base du protocole de l'étude et la création des sorties de données pertinentes avec le bon format (CDISC-SDTM, CDISC-ADaM pour l'analyse statistique, formats des outils de rapport). Cette dernière opération, souvent appelée "mapping des données", est particulièrement fastidieuse et longue. Elle consiste souvent à écrire des codes dans SAS ou d'autres technologies de manipulation des données.
Une solution consiste à promouvoir la standardisation des données, qui permet de réutiliser les paramètres de configuration et les codes, et donc de réduire la charge de travail liée aux tests de code nouvellement déployé. Cependant, la réutilisation de la configuration d'essais cliniques antérieurs peut ne pas être aussi facile qu'il n'y paraît. En effet, le copier-coller de conceptions d'essais précédents est un processus sujet à des erreurs lorsque les métadonnées sont dispersées dans plusieurs fichiers de paramètres. En fait, un gestionnaire de données peut même ne pas savoir où trouver les informations pertinentes sur les essais antérieurs.
Un référentiel de métadonnées cliniques (CMDR) est une plateforme logicielle capable de stocker et de mettre à jour/modifier les métadonnées des essais cliniques
La configuration des paramètres des systèmes EDC tels que les informations sur les visites, les formulaires et les questions (norme CDISC-ODM) ;
Les différentes versions de la norme CDISC-SDTM ou vos propres normes ;
La configuration du mapping des données.
Il permet également l'importation et l'exportation automatiques de métadonnées à partir de systèmes EDC compatibles (voir la figure ci-dessous).

En rassemblant toutes ces normes et en donnant accès aux métadonnées des études antérieures, les CMDR facilitent la réutilisation des configurations d'études antérieures. En effet, un gestionnaire de données cliniques peut facilement rechercher des métadonnées d'études similaires, en faire une copie, les modifier et les compléter, et demander l'approbation sans quitter la plateforme de CMDR. Certaines plateformes CMDR permettent même de visualiser les formulaires/questions, de sorte que la conception de l'étude peut être réalisée sans ouvrir le système EDC. En outre, comme les études de métadonnées précédentes ont déjà été validées, les platesformes CMDR atténuent également la charge de travail liée aux tests.
De plus, en fournissant un point de vérité unique dans toute votre organisation, les CMDR permettent une collaboration transparente entre les équipes et les pays. Comme la plupart des plateformes logicielles dans ce domaine, elles assurent également le contrôle de la traçabilité (qui a fait des mises à jour de quoi et quand), le contrôle de l'accès et le contrôle de versions des documents.
Tirer parti des techniques NLP pour promouvoir l'automatisation
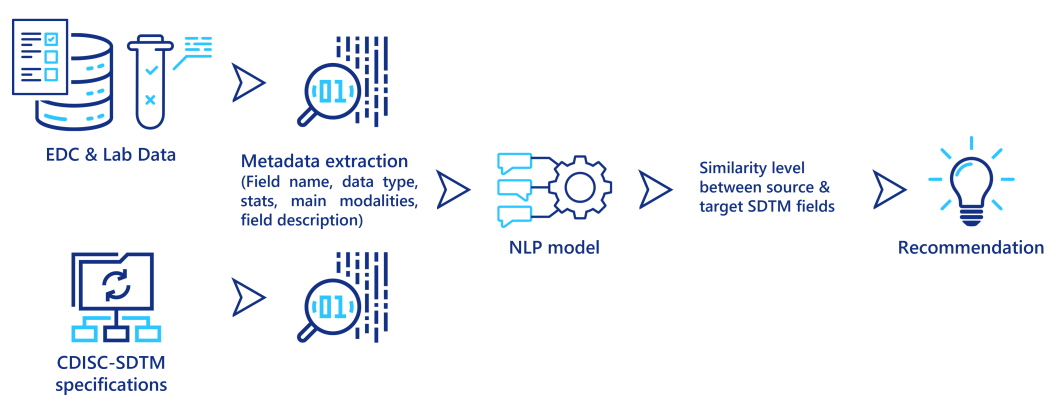
Les techniques de traitement du langage naturel (NLP) sont de plus en plus performantes pour extraire des informations pertinentes de données textuelles. En particulier, ces techniques sont capables d'identifier les similitudes entre deux descriptions textuelles différentes, afin de déterminer si elles décrivent toutes deux le même concept, objet ou entité. Elles peuvent être utilisées pour aider les gestionnaires de données cliniques lorsqu'ils mappent les données de leur système source (sortie EDC, données de laboratoire) à CDISC-SDTM. Certaines métadonnées sont d'abord extraites des données sources et des spécifications SDTM, puis pour chaque champ des données sources, un modèle NLP calcule un score de similarité avec les champs des normes SDTM et recommande les champs similaires aux gestionnaires de données (voir figure ci-dessous). Même si cette approche nécessite encore une validation finale de la part d'un gestionnaire de données cliniques, elle pourrait réduire considérablement le temps nécessaire pour effectuer ces tâches fastidieuses de mise en correspondance.
Une deuxième application des techniques NLP est la classification des documents. Ici, le type de document est automatiquement déduit de son contenu. Ceci est utile pour classer la quantité de documents générés au cours d'un essai clinique dans leur dossier correspondant dans le TMF, et ainsi assurer sa qualité. Il peut être particulièrement intéressant d'ingérer les données d'essais cliniques antérieurs stockées au format papier après les étapes de numérisation et de reconnaissance de caractères (OCR).
Une troisième technique de NLP est la reconnaissance d'entités nommées (NER). L'idée ici est de reconnaître/localiser des entités nommées (mot ou groupe de mots) et de les classer en catégories (nom, adresse, informations personnelles du patient, médicament, posologie, unités, critères d'éligibilité...). Les techniques de NER intègrent des taxonomies ou des ontologies spécifiques à un domaine et pourraient être utilisées pour extraire automatiquement des informations des protocoles d'étude. Cette technologie, ainsi que la promotion de modèles/standards de protocoles, peut ouvrir la voie à la configuration automatique de l'EDC à partir du protocole d'étude.
Ces techniques de traitement automatique des langues reposent principalement sur des modèles d'apprentissage automatique spécialement conçus pour le traitement des textes. Comme tout modèle, ses performances dépendent de la qualité et de la quantité de données utilisées pour l'entraîner. Au départ, il se peut que vous ne disposiez pas de suffisamment de données pour entraîner un bon modèle adapté à votre contexte, mais un modèle correctement intégré dans une plateforme logicielle pourrait être réentraîné chaque fois qu'un utilisateur corrige sa prédiction, et ainsi le modèle améliore son pouvoir de prédiction au fur et à mesure.
Même si le bénéfice réel de ces modèles doit être soigneusement évalué, ils sont très prometteurs pour soutenir et automatiser les tâches de gestion des données cliniques.
Comment tirer parti de ces innovations technologiques ?
Pour adopter correctement ces tendances technologiques, il est essentiel d'analyser les processus actuels et le stack technologique en place dans votre organisation, parallèlement à vos besoins actuels et futurs. Vous pourrez alors déterminer la meilleure façon d'avancer, qu'il s'agisse d'identifier les fournisseurs appropriés pour compléter votre stack existant ou de développer des preuves de concept pour évaluer la faisabilité et les impacts potentiels de ces technologies.
En s'appuyant sur notre expérience et nos capacités en tant que CRO et en tant qu'ESN, Keyrus peut vous aider à identifier vos besoins et vos contraintes et vous aider à façonner un plan d'action lors d'une étude de cadrage. Grâce à une main-d'œuvre polyvalente composée de gestionnaires de données cliniques, d'architectes de données, de scientifiques des données et de chefs de projets cliniques, nous pouvons vous accompagner pleinement dans votre transition technologique.
